Einführung
Die Entwicklung moderner Technologien zur Erfassung biologischer Prozesse führt zu einem unüberschaubar hohen Datenaufkommen. Diese Daten stammen sowohl aus Messungen als auch aus Simulationen von Prozessen einzelner Moleküle über die Expression von Genen bis hin zur Modellierung ganzer Populationen. Die Analyse solcher Daten ist besonders schwierig, da sie häufig fehlerbehaftet, heterogen sowie hochdimensional sind und daher ohne moderne Visualisierungs- und Analysetechniken nicht mehr auskommen würden.
Visuelle Analytik für Genexpressionsdaten
|
Speicher der Erbinformation aller Lebewesen auf der Erde ist die DNA, welche als lineare Abfolge von Nukleotiden vorliegt. Ein DNA-Abschnitt, der in ein Protein übersetzt werden kann, wird Gen genannt; die gesamte Erbinformation eines Organismus ist das Genom. Aufgrund moderner Hochdurchsatztechnologien sind mittlerweile zahlreiche Genome verschiedenster Organismen vollständig entziffert worden.
Unter Genexpression versteht man das Umschreiben (engl. transcription) der DNA in Boten-RNA (mRNA), die dann wiederum in Proteine übersetzt wird. Proteine führen die meisten Funktionen einer Zelle aus. Dabei ist die Genexpression ein hoch komplexer und genau regulierter Prozess, der es der Zelle erlaubt, dynamisch sowohl auf Umweltveränderungen als auch auf ihre eigenen wechselnden Bedürfnisse zu reagieren. Dieser Mechanismus agiert dabei sowohl als “an/aus”-Schalter – um zu kontrollieren, welche Gene in der Zelle exprimiert werden – wie auch als “Lautstärkeregler“, der den Grad der Genexpression erhöht oder verringert.
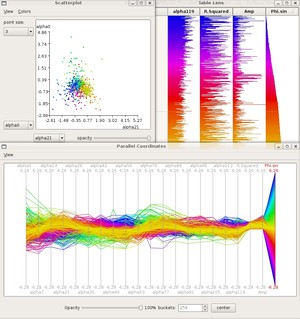
In den vergangenen Jahren wurden verschiedene Technologien entwickelt, die das parallele Messen der Expression von mRNA und Proteinen auf genomweiter Ebene erlauben. Bekannte Vertreter solcher Technologien sind Microarrays oder moderne Sequenzierverfahren, welche zur Analyse der Expression vieler Gene gleichzeitig verwendet werden können. Ziel solcher Messungen ist oft ein Vergleich der Expression zwischen zwei oder mehreren Zelltypen, beispielsweise zur Untersuchung gewebespezifischer Gene, der Genexpression in gesundem und krankem Gewebe, den Einfluss von Umweltveränderungen auf die Expression oder die Abhängigkeit der Genexpression vom Stadium des Zellzyklus. Als Ergebnis der Messungen erhält man folglich die Expression tausender Gene zu verschiedenen Konditionen, deren manuelle Analyse aufgrund des hohen Datenaufkommens nahezu unmöglich ist.
Visuelle Analytik für Genomweite Assoziationsstudien (GWAS)
|
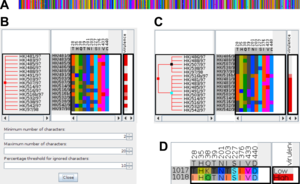
Auf der Suche nach Einzelnukleotid-Polymorphismen (SNPs), haben sind Genomweite Assoziationsstudien (GWAS) zu einer wichtigen Technik zur Identifikation von Assoziationen zwischen Genotyp und Phänotyp eines vielfältigen Sets Sequenz-basierter Daten etabliert. Genomweite Assoziationsstudien werden genutzt, um auftretende Variationen von Genen zwischen Individuen (Genotyp) und ihrer Assoziation mit einer Vielzahl komplexer Merkmale oder Krankheiten, wie z.B. Diabetes, Herzkrankheiten oder Arthritis, zu untersuchen. GWAS sind mittlerweile eine etablierte Methode zur Bestimmung von genetischen Risikofaktoren von Krankheiten, da sie neueste Techniken nutzen, welche eine schnelle und kosteneffiziente Analyse genetischer Unterschiede ermöglichen. Die großen Datenmengen, welche durch Genomweite Assoziationsstudien produziert werden, stellen eine große Herausforderung für die Datenanalyse und Visualisierung dar. Um Abhängigkeiten identifizieren und Zusammenhänge erkennen zu können, bedarf es einer adäquaten visuellen Repräsentation der Daten (mit geeignetem Mapping von Daten auf visuelle Attribute) und geeigneter Interaktion um die Sicht auf die Daten zu verändern. Letztere beinhaltet neben Fokus-und-Kontext-Techniken und dem Ein- und Ausblenden relevanter/uninteressanter Informationen auch das Aggregieren von Informationen, insofern sich diese (hierarchisch) sinnvoll gruppieren lassen.
Für die visuelle Analytik von genomweiten Assoziationsstudien wurde iHAT entwickelt.
Visuelle Analytik für biologische Netzwerke
Netzwerke spielen eine zentrale Rolle in der Erforschung von Organismen. Sie werden genutzt, um Prozesse biologischer Systeme zu repräsentieren und Interaktionen und Abhängigkeiten zwischen biologischen Entitäten wie z.B. Genen, Transkriptionen, Proteinen und Stoffwechselprodukten zu erfassen. Ein großes Anwendungsgebiet für Netzwerk-basierte Analyse und Visualisierung ist die System Biologie, welche ein umfassendes Verständnis bezüglich der Umformungsprozesse in Lebewesen anstrebt. Auf Grund des stetigen Wissenswachstums in den Biowissenschaften werden derartige Netzwerke immer komplexer und größer. Um dieses Problem der Komplexität anzugehen und die Analyse und Interpretation solcher komplizierten Netze von Interaktionen zu unterstützen, ist die Erstellung aussagekräftiger Visualisierungen von biologischen Netzwerken essentiell.
Die Visualisierung von biologischen Netzwerken wendet typischer Weise keine Standardtechniken zum Graphzeichnen ein, da diese in der Regel nicht die Konventionen der Biowissenschaftsgemeinschaft zum Graphzeichnen respektieren. Aus diesem Grund wurde der Entwicklung von automatischen Zeichenmethoden für biologische Netzwerke verstärkt Aufmerksam gewidmet mit dem Resultat dass diverse Layoutalgorithmen und eine Standardnotation (Systems Biology Graphical Notation) entwickelt wurden. Layout Algorithmen für biologische Graphen, müssen diverse Metainformationen, wie z.B. das Zellkompartiment in dem eine Reaktion stattfindet, in den Berechnungsprozess mit einbeziehen.
Weitere Herausforderungen für die Visualisierung von Graphen sind die Zeitkomponente und die Unsicherheit. Die Zeitkomente kann sich dabei auf die zeitliche Veränderung des Netzwerkes an sich (Reaktionen in Netzwerken sind mitunter zeitabhängig) oder bestimmter Eigenschaften der Entitäten, wie z.B. Expressionslevel, beziehen. Die Reaktionen welche in Netzwerken enthalten sind, treten oftmals nur zu einer bestimmten Wahrscheinlichkeit auf, was zu Unsicherheiten in den Netzwerken führt, welche es visuell hervorzuheben gilt. Das von uns entwickelte Tool iVUN kann dazu genutzt werden, Unsicherheiten geschätzer bzw. simulierter statischer aber auch dynamischer Parameter von biologischen Netzwerken zu analysieren.

